고차원 공간에서 유의미한 거리 매트릭스 이용하기
들어가기 전에
자연어 처리를 하다보면 단어나 형태소 등을 100차원 이상의 벡터로 나타내게 됩니다. 그리고 벡터화된 단어에 유클리디언 거리를 통해 단어간의 유사도를 비교하곤 합니다. (사실 gensim을 많이 이용하는데 여기서는 코사인 유사도를 이용하죠..) 아무래도 이런 방법들이 많이 쓰이다보니 별 생각없이 씁니다. 하지만 100차원 이상되는 고차원 공간에서 이와 같은 방법들이 제대로 된 유사도를 구해줄까요? On the Surprising Behavior of Distance Metrics in High Dimensional Space라는 논문에서는 그렇지 않다고 합니다. 그렇다면 어떤 방법을 써야 벡터간의 거리를 제대로 측정할 수 있을지 알아봅시다!
Norm
- norm은 () 입니다.
- 이때 k=1이면 Manhattan distance가 되는거고 k=2이면 uclidean distance가 됩니다. 그리고 k >= 3인 것보다 더 많이 쓰입니다.
- 빨간선, 파란선, 노란선은 Manhattan distance이고 초록선은 uclidean distance 입니다. Manhattan의 거리 구조가 그렇게 생겨서 이런 이름이 붙었다고 합니다.
k에 따른 최대값과 최소값의 차이
- Distance metric 를 사용했을 때 를 원점에서 가장 먼 point로, 를 원점에서 가장 가까운 point라고 해봅시다.
- 이런저런 증명을 해보면 는 의 rate로 증가한다고 합니다. 즉, k=1인 Manhattan distance metric은 무한대로 발산하고, k=2인 Euclidean distance metric은 상수값으로 수렴하며 다른 distance metric들은 0으로 수렴하게 됩니다.
- 그리고 k가 증가할 수록 수렴은 빨라집니다. 그렇다는 건 가장 가까운 점과 가장 먼 점 간의 차이가 거의 없어진다는 거겠죠.
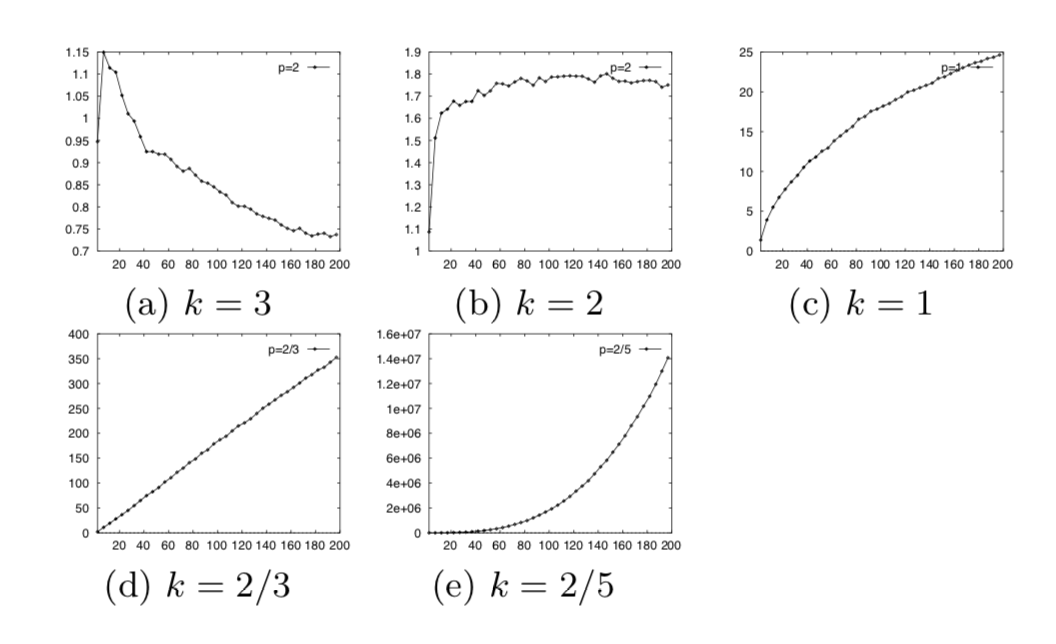
-
를 relative contrast라고 하고 라고 해봅시다. 그러면 는 Uclidean distance의 relative contrast이고 는 Manhattan distance의 relative contrast 이겠죠. 10개의 point가 있으며 uniform distribution에서 뽑았을 때, 를 계산해보면 아래와 같다고 합니다.

-
확실히 Manhattan distance metric이 Uclidean distance metric보다 relative contrast가 클 확률이 높습니다. 따라서 integral norm 중에는 k=1인 Manhattan distance metric이 가장 먼 점과 가장 가까운 점은 차이를 크게 하는데 최적의 선택이라고 할 수 있습니다.
-
아래 figure는 20차원인 point를 uniform 분포에서 뽑았을 때의 결과입니다. N은 point의 개수입니다.
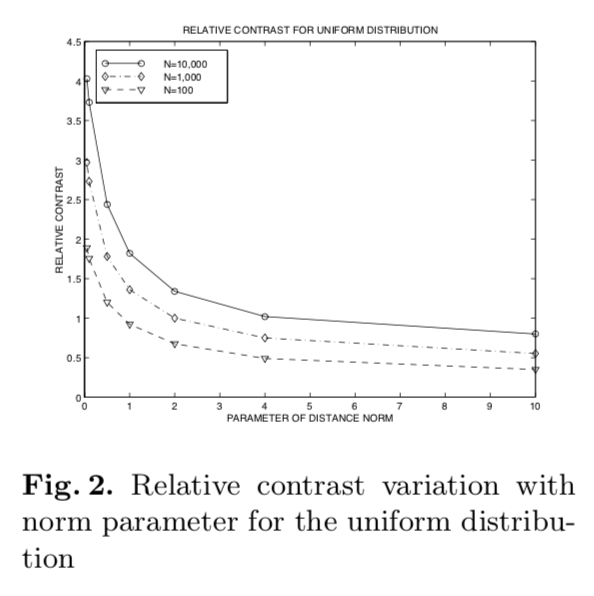
- k가 증가할수록 relative contrast가 감소하는 것을 알 수 있습니다.
- 흥미로운 점은 k < 1일 때, 즉 정수가 아닌 분수일 때 relative contrast가 훨씬 더 컸다는 것입니다. 그래서 논문 저자는 frational distance metrics가 integral distance metrics보다 더 나은 contrast를 준다고 합니다.
한 줄 요약
- norm에서 k가 정수일 때는 1이 좋고, 정수가 아니라면 분수가 좋다.
적용
-
한번 word embedding의 거리를 재는 데 적용해봅시다.
-
제가 확인해보고 싶었던 점은 두 가지 였습니다.
- k가 낮을 수록 높은 차원에서 유의미한 거리를 잴 수 있는가
- cosine similarity보다 좋은가
load embedding
from gensim.models import KeyedVectors
# path에서 gensim으로 학습 시켜놓은 word embedding을 가져옵니다.
wv = KeyedVectors.load({path})
# 행복이라는 명사의 topn 100개를 가져옵니다.
sample_words = [w for w,s in wv.most_similar('행복/NNG', topn=100)]
w2v = dict()
for w in sample_words:
w2v[w] = wv.get_vector(w)
w2v['행복/NNG'] = wv.get_vector('행복/NNG')
define distance function
import numpy as np
def get_l_k_dist(x, y, k):
return sum(abs(np.subtract(x,y)**k))**(1/k)
def get_euclidean_dist(query_word, w2v):
pairs = [(w, get_l_k_dist(w2v[query_word], w2v[w], 2)) for w in w2v]
sorted_pairs = sorted(pairs, key=lambda x: x[1])
sorted_pairs = sorted_pairs[1:]
return sorted_pairs
def get_manhattan_dist(query_word, w2v):
pairs = [(w, get_l_k_dist(w2v[query_word], w2v[w], 1)) for w in w2v]
sorted_pairs = sorted(pairs, key=lambda x: x[1])
sorted_pairs = sorted_pairs[1:]
return sorted_pairs
# get_l_k_dist의 k값에 분수를 넘겨주면 nan이 떠버리는데 왜 그런 걸까요...
def get_fractional_dist(query_word, w2v):
pairs = [(w, sum(abs(np.subtract(w2v[query_word], w2v[w])**1/2))**2) for w in w2v]
sorted_pairs = sorted(pairs, key=lambda x: x[1])
sorted_pairs = sorted_pairs[1:]
return sorted_pairs
compare similarity
# cosine similarity (gensim most_similar)
wv.most_similar('행복/NNG')
[('평안/NNG', 0.768101692199707),
('평온/NNG', 0.7442781925201416),
('편안/NNG', 0.7369856834411621),
('화목/NNG', 0.7297486066818237),
('불행/NNG', 0.7278854250907898),
('통쾌/NNG', 0.6954529881477356),
('우울/NNG', 0.6920645236968994),
('비참/NNG', 0.6906341314315796),
('무기력/NNG', 0.6847151517868042),
('암울/NNG', 0.678707480430603)]
# euclidean distance
euclidean_sims = get_euclidean_dist('행복/NNG', w2v)
euclidean_sims[:10]
[('평안/NNG', 34.26213724905869),
('평온/NNG', 34.89496449233225),
('불행/NNG', 35.80923190713129),
('화목/NNG', 35.871415005743486),
('편안/NNG', 36.10277110564432),
('통쾌/NNG', 37.60720168363042),
('우울/NNG', 38.0433546739748),
('비참/NNG', 38.10725296632495),
('무기력/NNG', 38.11499388672943),
('암울/NNG', 38.74589729467959)]
# manhattan distance
manhattan_sims = get_manhattan_dist('행복/NNG', w2v)
manhattan_sims[:10]
[('평안/NNG', 435.1311797052622),
('불행/NNG', 444.82760667800903),
('평온/NNG', 449.3698217496276),
('화목/NNG', 453.444153547287),
('편안/NNG', 457.6702497154474),
('통쾌/NNG', 476.65162378549576),
('우울/NNG', 487.55635057389736),
('무기력/NNG', 491.7380011975765),
('암울/NNG', 492.8903880119324),
('비참/NNG', 493.42741535604)]
# fractional_distance
fraction_sims = get_fractional_dist('행복/NNG', w2v)
fraction_sims[:10]
[('평안/NNG', 47334.785887923295),
('불행/NNG', 49467.89991572138),
('평온/NNG', 50483.30917482302),
('화목/NNG', 51402.900096553894),
('편안/NNG', 52365.51436865),
('통쾌/NNG', 56799.192614337444),
('우울/NNG', 59427.79874623428),
('무기력/NNG', 60451.565455446944),
('암울/NNG', 60735.233648638314),
('비참/NNG', 60867.653556235506)]
- 대체적으로 비슷한 결과가 나왔습니다. 그리고 k가 작아질수록 distance의 scale이 급격하게 증가합니다. Fractional distance(k=1/2)의 경우 sum값에 제곱을 해주기 때문에 scale이 매우 커졌습니다.
k의 영향
- 비참/NNG의 경우 k가 2보다 작아질 때 거리가 더 멀어졌습니다. cosine similarity와 euclidean distance에서는 같은 순위였기 때문에 방향이 큰 요인은 아닌 것 같습니다. 혹시 subtract를 했을 때 특이값이 영향을 준 걸 까요? 거리가 멀어졌던 비참/NNG와 거리가 가까워졌던 무기력/NNG subtract를 했을 때의 벡터 값이 어떻게 생겼는지 살펴봅시다.
v1 = wv.get_vector('행복/NNG')
v2 = wv.get_vector('비참/NNG')
v3 = wv.get_vector('무기력/NNG')
def get(v1, v2):
print('max', max(abs(np.subtract(v1,v2))))
print('avg', np.average(abs(np.subtract(v1,v2))))
print('var', np.var(abs(np.subtract(v1,v2))))
# 행복, 비참
get(v1, v2)
max 7.0177455
avg 1.9274508
var 1.957444
# 행복, 무기력
get(v1, v3)
max 7.1180973
avg 1.9208517
var 1.9851447
- 행복-비참이 행복-무기력보다 평균은 크지만 최대값과 분산 값이 낮음을 알 수 있습니다. 그렇다면 행복-무기력이 좀 더 특이한 값이 많지만 평균적으로 subtract값이 적은 벡터라고 볼 수 있고 k가 작아질 때 거리가 더 가까워졌으므로, k가 작아질 수록 특이값에 좀 더 robust하다고 볼 수 있습니다.
- 1번 질문에 대한 답으로 k가 낮을 수록 좀 더 벡터 간의 차이에 기반한 거리를 얻을 수 있다. 라고 결론 내릴 수 있겠습니다.
Cosine vs Norm
- 불행/NNG가 k가 2보다 작아질 때 거리가 더 가까워 졌습니다. cosine similarity로 할 때는 5번째로 가까운 단어였지만 norm을 사용할 때는 2번째, 3번째로 가까운 단어라고 나왔습니다. 아마, 불행/NNG는 행복/NNG와 비슷한 방향은 아니지만 비교적 가까운 거리에 위치하는 것 같습니다. cosine similarity는 거리가 얼마나 멀든지 두 벡터 사이의 cosine값을 이용하기 때문에 이든 이든 같은 방향에 있으므로 cosine similarity는 1입니다.
- 여기서 든 궁금증은 T-SNE를 이용해 클러스터를 유지하면서 2차원에 word embedding을 표현한다면 어떻게 될까였습니다. 그래서 T-SNE을 써서 word embedding을 2차원 공간상에 표현해봤습니다.
- 시각화 과정은 이곳을 참고했고 한글 폰트가 깨지는 문제점은 이곳을 참고했습니다.
from sklearn.manifold import TSNE
import matplotlib.pyplot as plt
import matplotlib.font_manager as fm
import pandas as pd
tsne = TSNE(n_components=2)
x_tsne = tsne.fit_transform(list(w2v.values()))
df = pd.DataFrame(x_tsne, index=w2v.keys(), columns=['x', 'y'])
path = '/usr/share/fonts/truetype/nanum/NanumGothic.ttf'
font_name = fm.FontProperties(fname=path, size=50).get_name()
fig = plt.figure()
fig.set_size_inches(40, 20)
ax = fig.add_subplot(1, 1, 1)
ax.scatter(df['x'], df['y'])
for word, pos in df.iterrows():
ax.annotate(word, pos, fontsize=30)
plt.show()
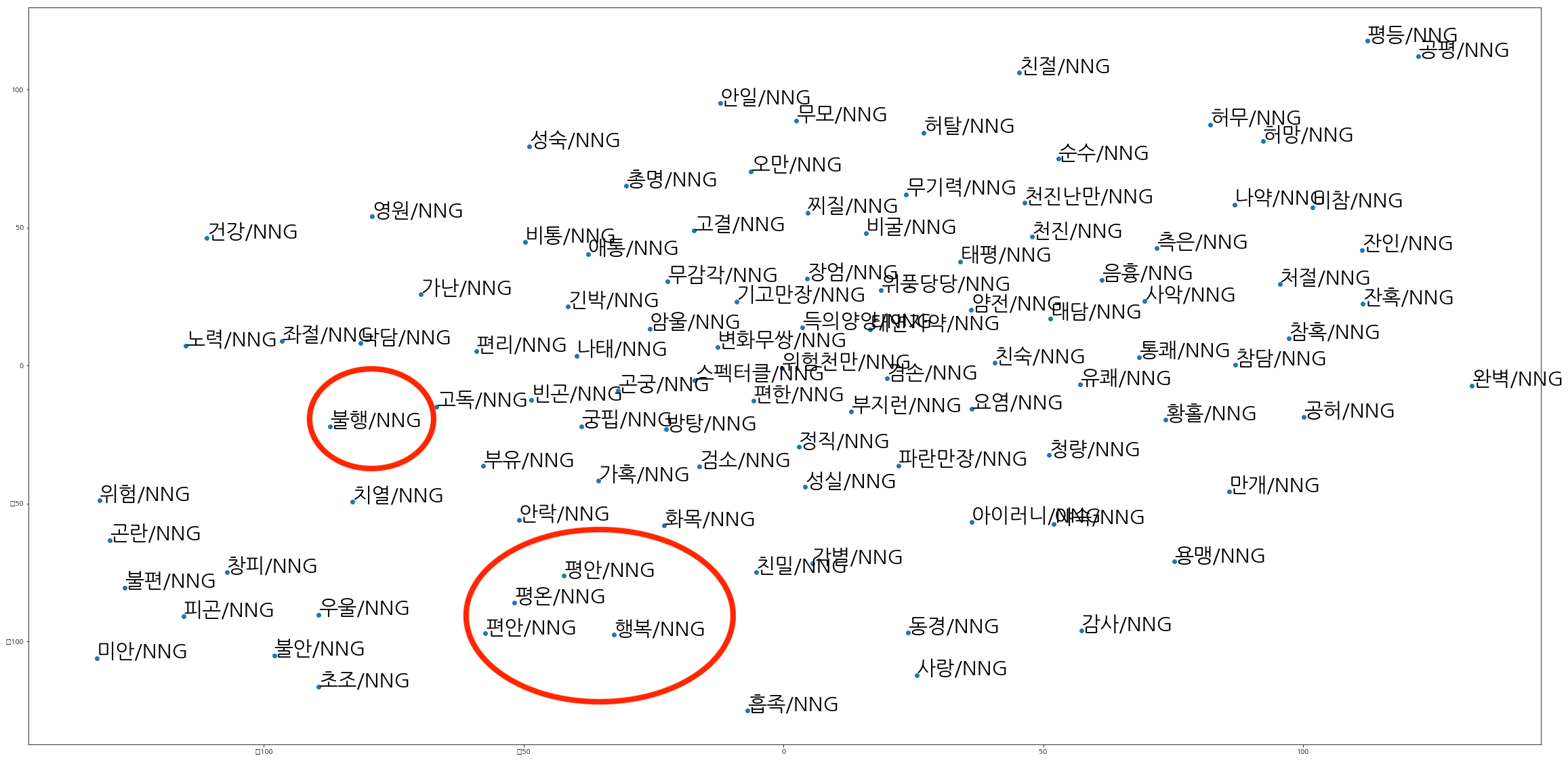
- 위와 같은 2차원 데이터를 볼 수 있습니다. 행복/NNG와 평안/NNG, 평온/NNG, 편안/NNG는 거리가 가까운 것을 확인할 수 있고 비교적 불행/NNG는 거리가 멉니다. 차원 축소 과정에서 불행은 행복과 멀어졌나 봅니다. T-SNE에서도 벡터 사이의 거리를 이용하는데 왜 이런 결과가 나오는지는 잘 모르겠습니다…
- 2번 질문에 대한 답은 정확히 내리기 어려울 것 같습니다. 하지만 T-SNE을 통해 살펴본 결과로는 어쩌면 cosine similarity가 더 나을지도 모르겠네요.
마무리
지금까지 norm에서 k가 norm에 미치는 영향을 알아보고 다른 거리 계산 방식인 cosine similarity와도 비교 해봤습니다. gensim의 most_similar를 이용하는 게 젤 무난할 것 같지만, 한번 norm도 이용해봐야겠습니다. 그리고 이때 euclidean distance 말고 manhattan이나 fractional distance를 사용해봐도 좋을 것 같습니다!


Leave a comment